在过去的十年中,染色质免疫共沉淀测序技术(ChIP-seq),一直是分析表观基因组和鉴定DNA相关蛋白重要结合位点的主要技术,其能够真实、完整地反映结合在DNA序列上的靶蛋白的调控信息。然而,ChIP-seq仍存在一些技术难点,在染色质制备和免疫沉淀两个关键步骤上,常常会造成样本丢失,导致ChIP-seq需要至少10,000个或数百万个细胞才能启动。

12月10日Nature Cell Biology期刊资讯,来自日本九州大学、东京工业大学、早稻田大学、东京大学和大阪大学的联合研究团队,开发出一种名为“染色质整合标记测序”(ChIL-seq)的新型表观基因组学分析方法,通过将免疫染色替代上述两个步骤,极大克服了样品丢失的问题。该方法无需免疫沉淀,可以利用极少量的细胞来分析DNA与蛋白质间的相互作用,为科学家研究稀有类型细胞及其他短缺的细胞样本提供新的机会。
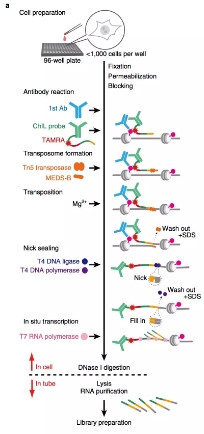
ChIL实验流程
在一系列评估实验中,研究人员成功证明,ChIL-seq技术仅使用100~1000个细胞就可以精确检测组蛋白修饰和DNA结合因子。利用ChIL-seq,研究团队还在单细胞水平上检测到与组蛋白标记相关的基因组区域,这也将有助于推动单细胞图谱的构建。此外,ChIL-seq技术还可以在细胞裂解前“放大”靶分子附近的基因组序列,这对于研究贴壁细胞等具有一定优势。这一新型技术可以捕获以前未检测到的表观基因组信息,揭示更多全新的生物标志物,进而推动精准医疗的进步。

ChIL反应示意模型
每种方法都具有其优势和局限性,ChIL-seq当然也不例外。ChIL-seq目前对异染色质区域的敏感性较低,并且可能耗时3~4天时间才能完成整个过程。但总的来说,凭借其精确性,ChIL-seq非常适用于单细胞领域的应用,其灵活性也意味着未来它可以与其他强大的测序技术相结合,发挥更大的作用。