靶向基因编辑猴、基因敲入食蟹猴、亨廷顿舞蹈症基因敲入猪,CRISPR-Cas9基因编辑系统已成功用于各种生物体,但有关编辑效率和脱靶效应的争议一直存在,很多人认为基因编辑的结果更像是随机的,无法预测,甚至可能产生意想不到的各种“bug”。结果不可预测、脱靶效应可谓是极大的问题。
12月14日,英国Francis Crick研究所成果发表在Molecular Cell上,他们发现CRISPR-Cas9基因编辑系列过程的背后有一种简单可预测的模式。通过对近1500个靶点进行了系统分析,发现不同位点间的基因编辑精确度(即特定indel的重现性)差异较大,某些靶点高度偏好一种indel,而另一些则显示出许多不常见的indel。
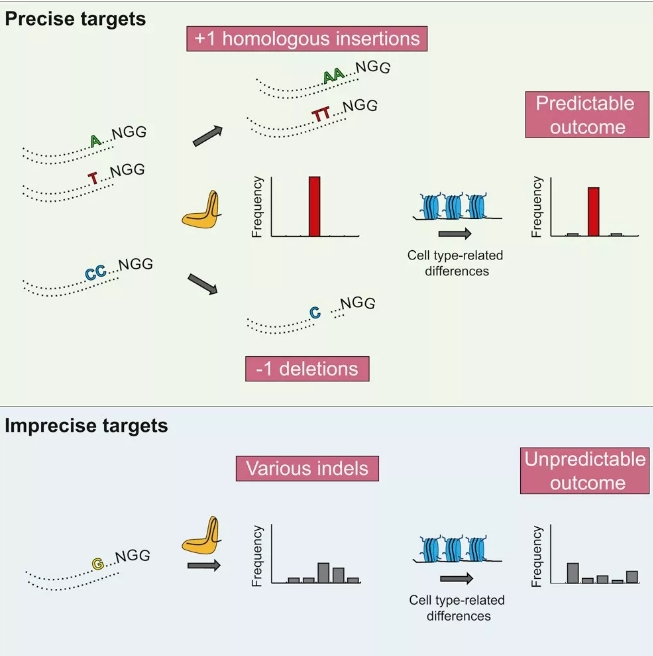
研究成果概述图
研究人员选择了450个核基因中的1491个位点,每个基因至少选择3个位点。结果共有1248个位点显示出可检测到的indel,每个靶点有1个到188个数量不等的indel,中位数为32。
在该研究中,有41个核苷酸位点显示出较长片段的缺失,44%和26%的靶点分别显示单核苷酸插入或缺失,是大多数靶点最常见的indel类型。根据聚类分析结果,研究人员将表现相似indel模式的靶点分为4种,即偏好小片段插入,小片段缺失,长片段缺失以及无明显偏好的位点。平均而言,给定位点最常见的indel频率为34.1%。
不同位点不仅对基因编辑的类型有不同的偏好,而且在数量上也有偏好。某些位点会出现多个不常出现的不同indel,而一些位点会主要显示某一indel。约五分之一的靶点至少有50%的机会能诱导特定的indel。

基因编辑精度与indel特征的关系
基因编辑精度与编辑效率、indel类型和indel大小有关,并且某些DNA序列对编辑精度有显著影响。如PAM序列上游的四个核苷酸会对编辑精确度产生影响,其中第四个单核苷酸的影响最大。仅一个“T”的存在,就能以一种可预测的方式,使该位点91%的概率引入插入,51%的概率修复。
有些位点很难成功诱导基因敲除,所有检测到的indel中有81%导致了移码突变,有3个位点对编码框内indel显示出强烈的偏好。该研究发现有助于我们检测不同位点的基因编辑精确度,可以大大提高临床应用的安全性。
